| |
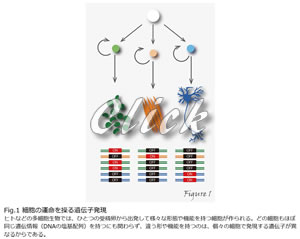
ヒトなど多細胞生物は、ひとつの受精卵から出発し、細胞の増殖と分化を経て個体が形成されます。完成された個体では、個々の細胞はそれぞれ特殊な形態や性質をもち、特化された役割を行使しています。例えばヒトの体は約60兆個の細胞から構成されていますが、どの細胞もほぼ同じ遺伝情報(DNAの塩基配列)を持つにも関わらず、違う形や機能を持つのはなぜでしょうか?それは、個々の細胞で発現する遺伝子が異なるからです(Figure1)。
この遺伝子発現の調節には、転写因子のみならず、DNAのメチル化やDNAと強く相互作用しているヒストンの翻訳後修飾によるエピジェネティックな制御が重要な働きをしていることが分かってきました。ヒストンの翻訳後修飾は、特に転写の抑制や活性化に働く多彩な修飾を受けることが知られています。 私たちは、ヒストンの翻訳後修飾や、転写を担うRNAポリメラーゼの生きた細胞内での動態を調べることで、細胞の機能や外部からのシグナルに応じて、遺伝情報が必要に応じて発現する仕組みを明らかにしようとしています。
【研究の背景】
「ヒストン」とは?
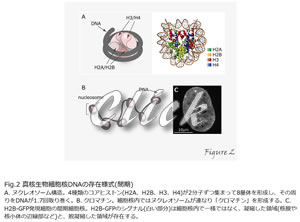
真核生物のDNAは、細胞核の中でヌクレオソームと呼ばれる構造を形成しています(Figure2)。
このヌクレオソームは、146~147塩基対のDNAがヒストン蛋白質8量体の周りを約2回(1.7回)取り巻く構造で、細胞核の中にDNAを収納するための基本単位です。ヌクレオソーム形成に働くヒストン蛋白質は、「コア」ヒストンと呼ばれ、H2A、H2B、H3、H4の4種類が2分子ずつ集まって8量体を形成します。細胞核の中には、このヌクレオソームが連なるクロマチンが非常に高密度で存在していますが、その分布は一様ではなく、一般に、転写されているクロマチン領域は脱凝縮して、逆に、転写が抑制されたクロマチン領域は凝縮しています。
ヒストンの翻訳後修飾とエピジェネティクス
クロマチン構造の形成は、DNAの収納にとどまらず、転写制御やDNAの複製、損傷修復などにも働きますが、ヒストンの翻訳後修飾は特にその制御に重要な役割を果たします(Kimura, 2013)。 ヒストンは、アセチル化、メチル化、リン酸化などの翻訳後修飾を受けます(Figure3)。
例えば、ヒストンH3のアセチル化は転写の活性化、H3の9番目や27番目のリジン残基のメチル化は転写の抑制に関与することが分かっています。これらのヒストン修飾を介して遺伝子発現が制御されることで、細胞分裂を経ても転写の抑制状態などが維持・継承されます。このようなDNA配列の変化を伴わずに起こる遺伝子発現抑制などの継承は「エピジェネティクス」と呼ばれています。また、ヒストン修飾やDNAメチル化などのエピジェネティクス情報を付加したゲノム情報は「エピゲノム」とも呼ばれます。ヒストン修飾によるエピジェネティクスの制御は酵母からヒトに至るまで見られる生物に普遍的な現象であり、その制御機構の解明は、再生医療やがん治療、幹細胞の形成など、細胞工学を使った多くの技術開発に結びつくと考えられます。
RNAポリメラーゼ
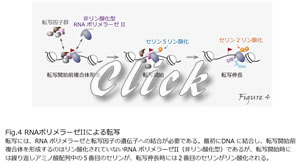
RNAポリメラーゼはDNAを鋳型としてRNAを合成する(つまり、転写を行う)蛋白質です。真核生物の細胞核には、3つのRNAポリメラーゼが存在します。RNAポリメラーゼIはリボソームRNA、RNAポリメラーゼIIは蛋白質をコードする遺伝子と一部の低分子RNA、RNAポリメラーゼIIIはtRNAなどの低分子RNAの転写にそれぞれ働きます。RNAポリメラーゼIIの転写には、特異的転写因子や基本転写因子により制御されています。RNAポリメラーゼIIが転写を行うためには、まず基本転写因子と共にプロモータ上で転写開始前複合体を形成し、転写開始、伸長へと至ります。この過程で、RNAポリメラーゼIIの最大サブユニットのC末端ドメインの繰り返しドメインのセリン残基がリン酸化されます。ヒトのRNAポリメラーゼIIのC末端ドメインは、Tyr-Ser-Pro-Thr-Ser-Pro-Serという7アミノが52回繰り返されていますが、転写開始時は5番目のSerが、転写伸長時には2番目のSerがリン酸化されます(Figure4)。
ヒストン修飾と転写活性化の生細胞イメージング
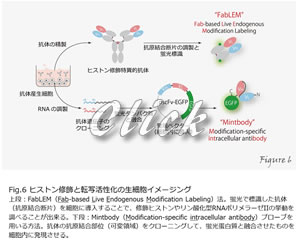
これまで説明してきた因子たちは、実際に細胞の中ではどのように活躍しているのでしょうか?蛍光蛋白質と融合させることで任意の蛋白質の局在を生きた細胞の中で観察することが出来るようになりました。しかし、全ての蛋白質分子が同じような機能を持つとは限らず、全蛋白質の局在を見るだけでは、蛋白質の機能を検出することはできません。ヒストンやRNAポリメラーゼIIのように、多くの蛋白質は、その機能に応じて様々な翻訳後修飾を受けるため、これらを指標として特別な機能を持つ分子のみを検出することができます。そのために、様々な修飾特異的抗体を開発してきました。特異的抗体を用いることで、免疫沈降や免疫染色により細胞内の修飾の局在を調べることが出来ます(Figure5)。
しかし、通常の免疫染色法では、化学固定した細胞を使用するため、時間変化を追うことができません。私たちは、蛍光で標識した抗体(抗原結合断片)を細胞に導入することで、修飾ヒストンやリン酸化型RNAポリメラーゼIIの挙動を調べることに成功しています(Kimura et al, 2010)。この方法はFabLEM(Fab-based live endogenous modification labeling)と名付けました(Hayashi-Takanaka et al, 2011)。また、抗体の抗原結合部位(可変領域)をクローニングして、蛍光蛋白質と融合させたものを発現できるような系も開発しました(Sato et al, 2013; 2016)。このような、特定の翻訳後修飾を受けた分子のみを選択的に生きた細胞や生体内で可視化する方法は、蛋白質の機能を調べる上で非常に有用であると考えられます(Figure6)。
 |
【研究内容】
ヒストンの翻訳後修飾は、遺伝子発現やDNA損傷修復などの染色体の機能の制御に非常に重要な役割を果たすことが分かってきました。私たちは、様々なヒストン修飾の意義と制御機構を明らかにするために、各種の修飾に対するモノクローナル抗体を作製してきました(Kimura et al, 2008)。この抗体を用いて、発生や分化、細胞周期の過程、あるいは外部刺激に対応したヒストン修飾の変化を調べています。ヒストン修飾は、個々の遺伝子領域で変動するほか、全体量の変化も起こります。ゲノム全域に渡るヒストン修飾の分布は、特異的抗体を用いたクロマチン免疫沈降により回収されたDNAの塩基配列を決定すること(いわゆるChIP-seq法)で調べることができます。また、私たちは、少数の細胞のヒストン修飾の分布を調べる方法を開発しています(Harada et al, 2019)。一方、ヒストン修飾の細胞内局在や相対的存在量は、化学固定した細胞を用いた免疫染色により解析できますが、私たちは、 生きた細胞や生体内でヒストン修飾や転写の動態を調べる方法を開発してきました。これらの方法により、染色体分配に関わるヒストンH3のリン酸化の制御が正常細胞 とがん化した細胞で異なることや転写制御に関わるヒストンH3のアセチル化へのヒストン脱アセチル化酵素阻害剤の影響などを明らかにしてきました(Hayashi-Takanaka et al, 2009; 2011; Sato et al, 2013)。また、H3のアセチル化が転写の開始から伸長に至る過程を促進することも明らかにしました(Stasevich et al, 2014)。現在、細胞が分化する過程や外部刺激に反応して起こる種々のヒストン修飾の変化を調べています(Sato et al, 2019)。
【参考文献】
Sato Y, Hilbert L, Oda H, Wan Y, Heddleston JM, Chew TL, Zaburdaev V, Keller P, Lionnet T, Vastenhouw N, Kimura H. (2019), an Y, Heddleston JM, Chew TL, Zaburdaev V, Keller P, Lionnet T, Vastenhouw N, Kimura H. Histone H3K27 acetylation precedes active transcription during zebrafish zygotic genome activation as revealed by live-cell analysis. Development 146, dev179127.
Harada A, Maehara K, Handa T, Arimura Y, Nogami J, Hayashi-Takanaka Y, Shirahige K, Kurumizaka H, Kimura H, Ohkawa Y. (2019) A chromatin integration labelling method enables epigenomic profiling with lower input. NCB 21, 287-296.
Sato Y, Kujirai T, Arai R, Asakawa H, Ohtsuki C, Horikoshi N, Yamagata K, Ueda J, Nagase T, Haraguchi T, Hiraoka Y, Kimura A, Kurumizaka H, Kimura H. (2016) A genetically encoded probe for live-cell imaging of H4K20 monomethylation. JMB 428, 3885-3902.
Stasevich TJ, Hayashi-Takanaka Y, Sato Y, Maehara K, Ohkawa Y, Sakata-Sogawa K, Tokunaga M, Nagase T, Nozaki N, McNally JG, and Kimura H. (2014). Regulation of RNA II activation by histone acetylation in single living cells. Nature 516, 272-275.
Sato Y, Mukai M, Ueda J, Muraki M, Stasevich TJ, Horikoshi N, Kujirai T, Kita H, Kimura T, Hira S, Okada Y, Hayashi-Takanaka Y, Obuse C, Kurumizaka H, Kawahara A, Yamagata K, Nozaki N, and Kimura H. (2013). Genetically encoded system to track histone modification in vivo. Sci Rep 3, 2436.
Kimura H. (2013). Histone modification for human epigenome analysis. J Hum Genet 58, 439-445.
Hayashi-Takanaka Y, Yamagata K, Wakayama T, Stasevich TJ, Kainuma T, Tsurimoto T, Tachibana M, Shinkai Y, Kurumizaka H, Nozaki N, and Kimura H. (2011). Tracking epigenetic histone modifications in single cells using Fab-based live endogenous modification labeling. Nucleic Acids Res 39, 6475-6488.
Kimura H, Hayashi-Takanaka Y, and Yamagata K. (2010). Visualization of DNA methylation and histone modifications in living cells. Curr Opin Cell Biol 22, 412-418.
Hayashi-Takanaka Y, Yamagata K, Nozaki N, and Kimura H. (2009). Visualizing histone modifications in living cells: spatiotemporal dynamics of H3 phosphorylation during interphase. J Cell Biol 187, 781-790.
Kimura H, Hayashi-Takanaka Y, Goto Y, Takizawa N, and Nozaki N. (2008). The organization of histone H3 modifications as revealed by a panel of specific monoclonal antibodies. Cell Struct Funct 33, 61-73.

|